ladyLein startpagina
0. R install & library 1. R start 2. Vectoren (een rij waarden) 3. Matrixen (tabellen) 4. Statistieken (min, max, gemiddelde, standaard defiatie) 5. Factoren (-categorieën/typen) 6. Data frames (wat is een dataframe, inlezen txt en csv bestanden) 7. Correlatie in de grafiek 8. Plot 9. GGplot2 (lijn, staaf, taart) 10. Functies (o.a.if else, for, while, string, datum) 11. SQL / dplyr (gegevens ophalen uit tabellen) 12. WAR meetkastjes en KNMI - Locaties meetkastjes - Data één meetkastje - Meerdere meetkastjes +tijdreeks - Waar welke meetkastjes - KNMI data - WAR met KNMI data - Analyse meetkastjes - Waarnemingen 13. Qgis gegevens op de kaart zetten |
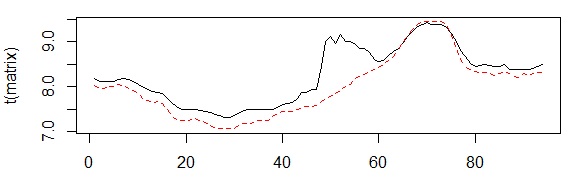
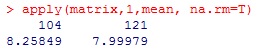
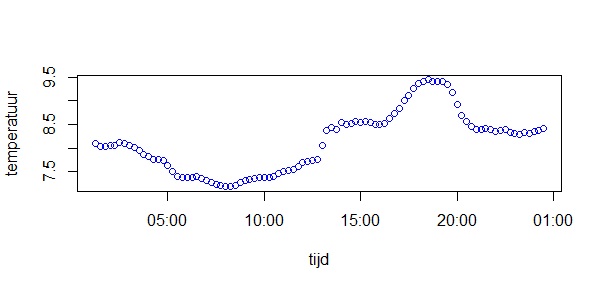
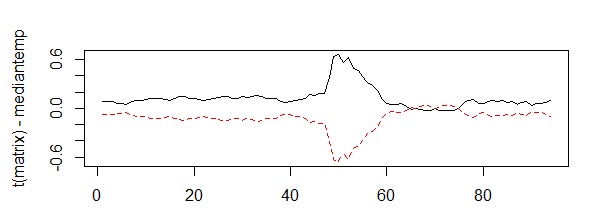
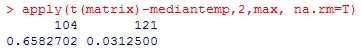
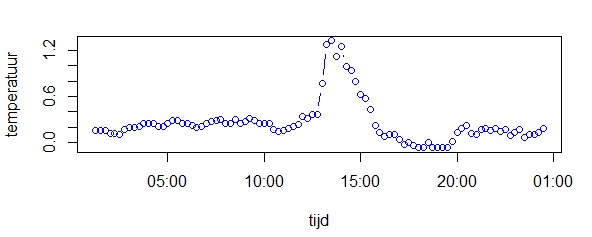
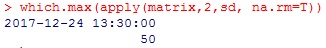
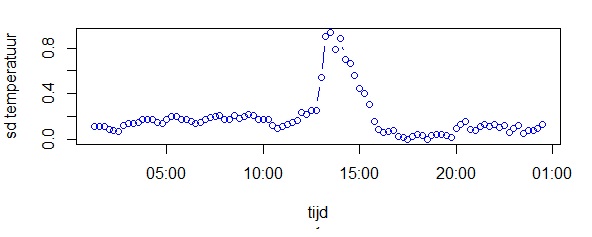
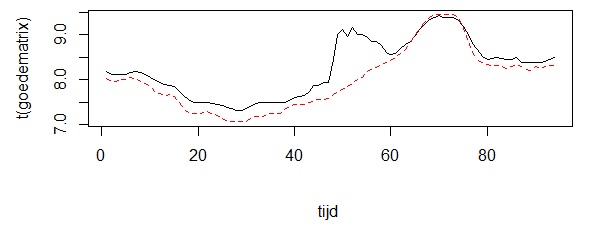
Analyse meetkastjes dataHaal de data op Zet de tijden goed, want ze zenden niet allemaal tegelijk hun data door Doe de analyses Haal de data van meerdere meetkastjes op# maak een variabele genaamd 'start', met het beginmoment van je grafiekstart<-"2017-10-20,00:00" # maak een variabele genaamd 'eind', met het eindmoment van je grafiek end<-"2018-01-15,23:59" # maak variabele 'ids', met het nummer van de sensor die je wilt bekijken # voorbeelden: "13", "13,14", "1-50", "1,2,10-20", etc. ids<-"1-50" ids<-"121,104" ids<-"11,14,19,26,31,37,41,47" (windroos) ids<-"67,60,74,22,10" (centrum, west, zuid, noord, oost) # haal de data op van de server van Meet je Stad data<-read.table(paste("http://meetjestad.net/data? type=sensors&start=",start,"&end=",end,"&ids=",ids,"&format=csv", sep=""), sep="\t", header=T, fill=T) # op de server zijn alle metingen opgeslagen in Universal Time Code. Laten we deze globale tijd omrekenen naar onze lokale tijd ts<-as.POSIXct(data$timestamp, origin="1970-01-01", tz="UTC") data$localtime<-as.POSIXct(ts, "CET") attributes(data$localtime)$tzone <- "CET" # zoek de hoogste en de laagste temperatuur van de opgehaalde datareeks # we gebruiken het 5e en 95e percentiel van de meetwaarden, en trekken daar 1 graad of of tellen het erbij ylow<-quantile(data$temperature,0.05)-1 yhigh<-quantile(data$temperature,0.95)+1 # maak een grafiek van de data (lijnen) ggplot(data=data, aes(localtime,temperature, colour = as.factor(id))) + geom_line(aes(group = as.factor(id)) ) + ylim(ylow,yhigh) 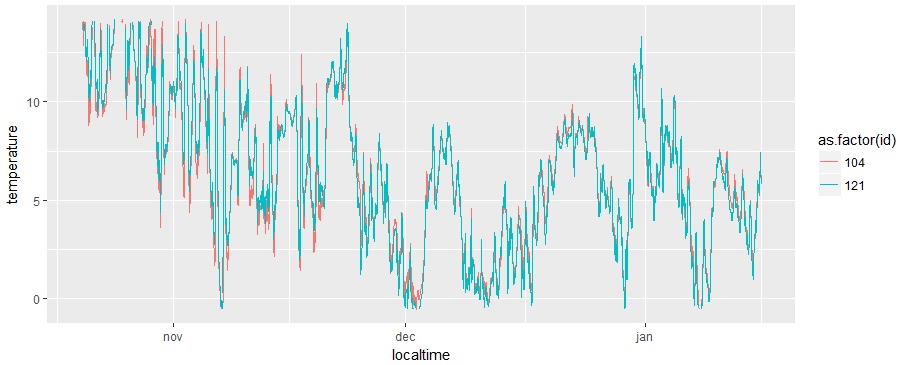 Zet de tijden goed# Maak een vector met de vroegste en laatste tijdtsrange <- range(data$localtime) # rond deze start- en eindtijd af naar het dichtstbijzijnde kwartier startkwartier<-as.POSIXlt(round(as.double(tsrange[1])/(15*60))*(15*60),origin=(as.POSIXlt('1970-01-01'))) eindkwartier<-as.POSIXlt(round(as.double(tsrange[2])/(15*60))*(15*60),origin=(as.POSIXlt('1970-01-01'))) # maak een reeks van het beginkwartier tot het eindkwartier in stappen van 15 minuten tssequence <- seq(startkwartier+15*60,eindkwartier-15*60, by=15*60) # maak een data frame met een lege matrix voor alle stations en kwartieren matrix<-data.frame(matrix(,nrow=length(levels(factor(data$id))),ncol=length(tssequence) ) ) # vul de matrix # van iedere reeks metingen van een station, # berekenen we de lineaire interpolartie naar de kwartier tijden j<-0 for (i in as.numeric(levels(factor(data$id))) ) { tree <- subset(data, id==i) ltime <- tree$localtime if (length(ltime) > 1) { j<-j+1 interpol<-approx( as.numeric(ltime),tree$temperature, xout=as.numeric(tssequence), rule=1, method = "linear", ties=mean) matrix[j,] <- c(interpol$y) } } # geeft de kolommen en rijen namen colnames(matrix)<-tssequence rownames(matrix)<-levels(factor(data$id)) ja ja .... (ik doe ook maar kopieren en plakken) Doe de analyses
Terug naar top |